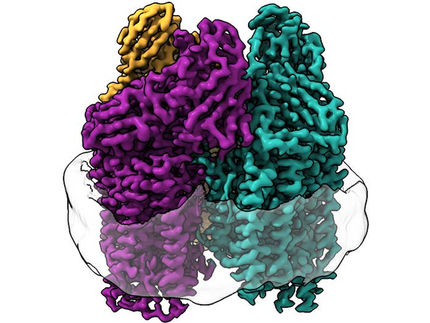
¿Cómo convierten los motores moleculares la energía química en trabajo mecánico?
Los investigadores revelan la estructura y función de una compleja micromáquina
Los motores moleculares son dispositivos complejos compuestos por muchas partes diferentes que consumen energía para realizar diversas actividades celulares. En resumen, las máquinas moleculares transforman la energía en trabajo útil. La comprensión de los aspectos mecánicos que subyacen a estos motores empieza por generar una descripción detallada de su arquitectura general y su organización atómica. Sin embargo, para descubrir los mecanismos centrales que dan energía a estos motores es esencial descifrar toda la dinámica molecular en detalle atómico.
Representación artística de la unión Holliday y los motores RuvB.
CSSB, Nicola Graf
Ahora, el equipo de investigación de Thomas C. Marlovits, del Centro de Biología de Sistemas Estructurales CSSB del DESY y del Centro Médico Universitario Hamburgo-Eppendorf (UKE) de Hamburgo, revela la arquitectura, el ciclo funcional completo y el mecanismo de dicho motor molecular: Informan en la revista Nature de cómo un "complejo de migración de ramas RuvAB" convierte la energía química en trabajo mecánico para llevar a cabo la recombinación y la reparación del ADN.
La recombinación del ADN es uno de los procesos biológicos más fundamentales en los organismos vivos. Es el proceso por el que los cromosomas "intercambian" ADN, ya sea para generar diversidad genética, creando nueva descendencia, o para mantener la integridad genética, reparando roturas en los cromosomas existentes. Durante la recombinación del ADN, cuatro brazos de ADN se separan de sus formaciones de doble hélice y se unen en una intersección conocida como unión Holliday. En este punto, los brazos de ADN intercambian cadenas en un proceso denominado migración activa de ramas.
La energía esencial necesaria para que se produzca esta migración de rama proviene de una maquinaria molecular que los científicos han etiquetado como el complejo de migración de rama RuvAB. Este complejo se reúne alrededor de la unión Holliday y está formado por dos motores etiquetados como RuvB AAA+ ATPasas, que alimentan la reacción, y un estator RuvA. El equipo de investigación ha proporcionado ahora un intrincado esquema que explica cómo los motores RuvB AAA+ trabajan bajo la regulación de la proteína RuvA para realizar el movimiento sincronizado del ADN.
Las migraciones activas de las ramas energizadas por la molécula del motor RuvB AAA+ son muy rápidas y altamente dinámicas. Para determinar los pasos individuales de este proceso, los científicos utilizaron la criomicroscopía electrónica con resolución temporal para observar la maquinaria del motor en cámara lenta. "Básicamente, dotamos al motor RuvB AAA+ de un combustible de combustión más lenta que nos permitió captar las reacciones bioquímicas a medida que se producían", explica Marlovits.
El científico capturó más de diez millones de imágenes de la maquinaria del motor interactuando con la unión Holliday. Jiri Wald (CSSB, UKE y parte del programa de doctorado del BioCentro de Viena), primer autor del artículo, peinó la inmensa cantidad de datos y clasificó cuidadosamente los sutiles cambios que se producían en cada imagen. Utilizando las instalaciones de computación de alto rendimiento del DESY, los científicos fueron capaces de unir todas las piezas del rompecabezas para generar una película de alta resolución que detallaba el funcionamiento del complejo RuvAB a escala molecular.
"Pudimos visualizar siete estados distintos del motor y demostrar cómo los elementos interconectados funcionan juntos de forma cíclica", explica Wald. "También demostramos que el motor RuvB convierte la energía en un movimiento de palanca que genera la fuerza que impulsa la migración de las ramas. Nos sorprendió el descubrimiento de que los motores utilizan un mecanismo básico de palanca para mover el sustrato de ADN. En general, el mecanismo secuencial, la coordinación y la forma de generar fuerza del motor RuvAB comparten similitudes conceptuales con los motores de combustión".
Los motores AAA+ se utilizan a menudo en otros sistemas biológicos, como el transporte de proteínas, por lo que este modelo detallado del motor RuvB AAA+ puede servir de modelo para motores moleculares similares. "Entendemos cómo funciona el motor y ahora podemos ponerlo en otro sistema con algunas adaptaciones menores", explica Marlovits. "En esencia, estamos presentando los principios básicos de los motores AAA+".
El trabajo futuro del grupo de Marlovits explorará formas de interferir en la función de los motores AAA+. Esto podría sentar las bases para el desarrollo de una nueva generación de fármacos, que interrumpirían los mecanismos de dicho motor en los patógenos y, por tanto, detendrían la propagación de la infección. "Nos entusiasma explorar las posibilidades que existen ahora que tenemos un plano del motor RuvB AAA+", señala Wald.
En esta investigación han colaborado científicos de CSSB, UKE, el Instituto de Biotecnología Molecular, el Instituto de Investigación de Patología Molecular, ambos en Viena (Austria), y el DESY.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.