los "diccionarios" hacen accesibles los datos basados en la fluorescencia para los modelos de estructura integradora y su dinámica
estructuras tridimensionales de biomoléculas
Un equipo de investigación de Alemania y EE.UU. dirigido por la Universidad Heinrich Heine de Düsseldorf (HHU) ha desarrollado una descripción de datos que puede proporcionar resultados de mediciones de fluorescencia para la modelización estructural y dinámica de grandes biomoléculas. Los autores explican en la revista científica Nature Methods que, por primera vez, otros investigadores pueden acceder a modelos estructurales integradores basados en fluorescencia y a su dinámica a través de bases de datos. Esto proporciona datos de entrenamiento basados en experimentos para la próxima generación de herramientas de IA para el modelado dinámico de estructuras.
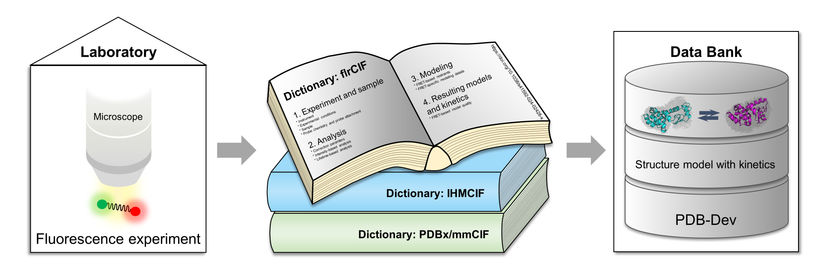
Los datos de los experimentos de fluorescencia se procesan con ayuda de "diccionarios" y se ponen a disposición junto con modelos de estructura integradora en una base de datos.
HHU / Christian Hanke
Las proteínas y los ácidos nucleicos son los componentes básicos de la vida en todos los organismos. Estas biomoléculas están formadas por muchos componentes individuales, como los aminoácidos en el caso de las proteínas. Cuando se ensamblan en las células, las biomoléculas forman estructuras tridimensionales complejas. Su forma específica viene determinada por la configuración y las fuerzas entre los componentes. Sin embargo, para comprender la función de las biomoléculas, es importante tener en cuenta no sólo su estructura tridimensional, sino también el número de estados estructurales diferentes y la dinámica de intercambio entre ellos.
Durante mucho tiempo, determinar la estructura tridimensional de las biomoléculas mediante métodos biofísicos clásicos ha sido muy complejo y ha llevado mucho tiempo. Para simplificar y sistematizar este trabajo paso a paso, todas estas estructuras tridimensionales se recogen desde 1971 en el "Protein Data Bank" (PDB). Estas más de 220.000 estructuras son utilizadas por herramientas basadas en IA como "AlphaFold" -por cuyo desarrollo se ha concedido este año el Premio Nobel de Química- como datos de entrenamiento para redes neuronales. Los sistemas de IA ya hacen buenas predicciones de estructuras biomoleculares. Sin embargo, en la actualidad estas herramientas son incapaces de predecir la dinámica por falta de datos experimentales.
Por eso es importante utilizar métodos experimentales potentes, como la espectroscopia de fluorescencia, que proporcionan información combinada sobre la dinámica y la estructura de biomoléculas complejas. En los experimentos de fluorescencia, las partes específicas de interés de las biomoléculas se marcan con pequeñas moléculas de colorante que se iluminan cuando se excitan externamente y revelan así sus posiciones. Los modelos integradores combinan estos datos experimentales con métodos computacionales para lograr una mayor resolución estructural y tener en cuenta la dinámica.
El Dr. Christian Hanke, postdoctorando en el Instituto de Química Física Molecular de la HHU y primer autor del artículo publicado en Nature Methods, subraya: "Los experimentos de fluorescencia proporcionan información detallada, lo que los convierte en una excelente fuente de datos para la modelización integradora. Sin embargo, para utilizar plenamente esta riqueza de información, debe hacerse accesible y utilizable por una amplia comunidad científica".
En la publicación, el equipo de investigadores de la HHU, la Universidad Estatal Rutgers de Nueva Jersey y la Universidad de California en San Francisco presenta una descripción normalizada de los datos en forma de tres "diccionarios" enlazados que se organizan en una biblioteca común. El Prof. Dr. Claus Seidel, de la HHU, uno de los dos autores correspondientes: "Este principio organizativo con diccionarios combinados permite a los investigadores almacenar por primera vez modelos estructurales integradores basados en datos de fluorescencia junto con información cinética. Al mismo tiempo, las definiciones generales también pueden ser utilizadas por otros métodos para documentar propiedades dinámicas de biomoléculas junto a su estructura en la base de datos."
Este enfoque es necesario para vincular los modelos estructurales estáticos con el paisaje energético subyacente, es decir, las diferencias de energía entre las distintas disposiciones tridimensionales de los bloques de construcción dentro de la biomolécula. El profesor Seidel: "Esta información permite desarrollar y entrenar la próxima generación de programas basados en IA para predecir estructuras biomoleculares dinámicas. Aquí, los datos obtenidos a partir de experimentos de fluorescencia sobre dinámicas funcionalmente relevantes pueden suponer una contribución muy importante."
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.