Archivo de datos con moléculas
Lectura más eficaz de polímeros sintéticos como almacenamiento de datos
Anuncios
Cada vez es necesario almacenar más datos, a menudo a largo plazo. Los polímeros sintéticos son una alternativa a los medios de almacenamiento convencionales, ya que retienen la información almacenada con unos requisitos de espacio y energía significativamente menores. Sin embargo, la lectura de datos por espectrometría de masas limita la longitud y, por tanto, la capacidad de almacenamiento de las cadenas poliméricas individuales. En la revista Angewandte Chemie, un equipo de investigadores presenta un nuevo enfoque que supera esta limitación y permite acceder directamente a los bits de interés sin necesidad de leer toda la cadena.
Wiley-VCH
Los datos se generan a diario, ya sea en el contexto de transacciones comerciales, control de procesos, garantía de calidad o trazabilidad de lotes de producción. Archivar estos datos durante décadas requiere mucho espacio, pero también energía. Las macromoléculas con una secuencia definida, como el ADN y los polímeros sintéticos, son una alternativa interesante, sobre todo para archivar a largo plazo grandes cantidades de datos a los que sólo es necesario acceder con poca frecuencia.
En comparación con el ADN, los polímeros sintéticos ofrecen ventajas: síntesis sencilla, mayor densidad de almacenamiento y estabilidad en condiciones adversas. La desventaja es que la información codificada en los polímeros se lee mediante espectrometría de masas (EM) o secuenciación de masas en tándem (EM2). Para ello, las moléculas no deben ser demasiado grandes, lo que limita mucho la capacidad de almacenamiento por cadena. Además, toda la cadena se lee módulo por módulo; no es posible acceder directamente a los bits de interés, como si hubiera que leer un libro entero en lugar de mirar la página correspondiente. En cambio, las cadenas largas de ADN pueden descomponerse en fragmentos de longitud aleatoria, secuenciarse individualmente y reconstruirse computacionalmente en la secuencia global.
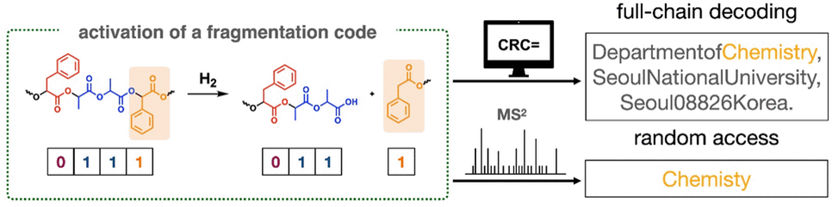
Kyoung Taek Kim y su equipo del Departamento de Química de la Universidad Nacional de Seúl (Rep. de Corea) desarrollaron un nuevo método con el que se pueden leer de forma eficiente cadenas de polímeros sintéticos muy largas, cuyos pesos moleculares superan considerablemente el límite analítico de la EM o la EM2. Como ejemplo, codificaron la dirección de su universidad en un código ASCII y lo tradujeron -junto con un código de detección de errores (CRC, un método habitual para comprobar la integridad de los datos)- a un código binario, es decir, una secuencia de 1 y 0. Almacenaron la información de 512 bits generada de este modo en una cadena polimérica formada por dos monómeros diferentes: Ácido láctico con código 1 y ácido fenil-láctico con código 0. También incorporaron códigos de fragmentación que contenían ácido mandélico en puntos irregulares. Tras la activación química, las cadenas se dividen allí, en el ejemplo en 18 fragmentos de distintos tamaños, que pueden descodificarse individualmente mediante secuenciación MS2.
Un software especialmente desarrollado identifica primero los fragmentos basándose en su masa y sus grupos finales a partir de los espectros MS. Durante la MS2, los iones moleculares que ya se han medido se "rompen" aún más y los fragmentos se analizan de nuevo. Los fragmentos pueden secuenciarse en función de sus diferencias de masa. Utilizando los códigos de detección de errores CRC, el software reconstruye la secuencia de toda la cadena. Así se supera la limitación de longitud de las cadenas poliméricas.
El equipo también consiguió leer bits de interés sin secuenciar toda la cadena polimérica (acceso aleatorio), por ejemplo, la palabra "Química" del código de la dirección. Teniendo en cuenta que todas las partes de la dirección están separadas por comas y dispuestas en un orden específico (departamento, institución, ciudad, código postal, país), fue posible acotar el lugar donde se almacena la información buscada dentro de la cadena y secuenciar sólo los fragmentos relevantes.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Alemán se puede encontrar aquí.